Introduction
Depuis plus d'un an maintenant, l'attention du monde s'est portée sur Politique Covid-19, infection respiratoire causée par le nouveau Virus Corona SARS-CoV-2, et à la pandémie qui en a résulté.
Les données épidémiologiques du moment disent que : le SARS-CoV-2 est présent dans plus de 200 pays du monde, environ 19 millions de personnes sont tombées malades avec COVID-113 dans le monde (février 2021) et, parmi celles-ci, pas de 2,5 millions décédés.
Le SRAS-CoV-2 est un virus qui affecte principalement les voies respiratoires, provoquant des symptômes tels que toux, rhume, fièvre et, dans les cas les plus graves, des difficultés respiratoires ; parfois, cependant, il peut également induire une inflammation systémique, provoquant une septicémie, une insuffisance cardiaque et un dysfonctionnement de plusieurs organes.
L'infection par le SRAS-CoV-2 est particulièrement dangereuse pour les personnes de plus de 60 ans, pour les personnes atteintes de maladies chroniques (ex : diabète, maladie coronarienne) et pour les personnes sous thérapie avec des médicaments dépresseurs du système immunitaire (ex : chimiothérapie, immunosuppresseurs).
Cet article vise à analyser la structure, le génome et les protéines du SARS-CoV-2, et à fournir des informations fondamentales liées à la pathogenèse du virus.
Pour plus d'informations : SARS-CoV-2 : comment reconnaître les premiers symptômes et que faireStructure du SRAS-CoV-2
Comment il est fabriqué SARS-CoV-2 : Structure, génome et protéines
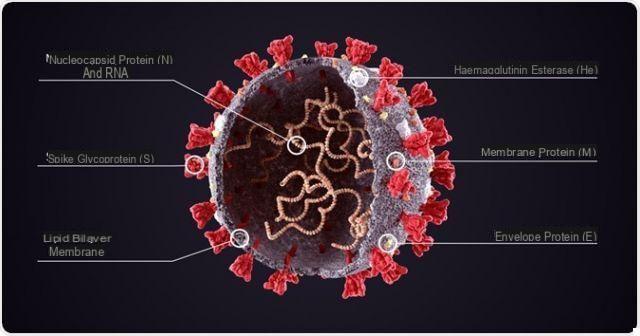
Exemple de bêta-coronavirus, le SARS-CoV-2 est un virus à ARN simple brin positif, équipé de péricapside (l'enveloppe).
La péricapside est une sorte d'enveloppe placée autour de la capside de certains virus ; il est composé de phospholipides et de glycoprotéines.
Le SARS-CoV-2 possède un génome de 29.881 XNUMX bases azotées, quels codes pour 9.860 acides aminés.
Ce génome se décompose en gènes pour protéines structurelles et des gènes pour protéines non structurales.
Les gènes des protéines de structure codent pour pic de protéines (abrégé en S), la protéine de la péricapside (abrégé en E, de l'enveloppe), le protéine membranaire (abrégé en M) et le protéine de nucléocapside (abrégé en N).
Comme son nom l'indique, les protéines structurelles se combinent pour former la structure du SARS-CoV-2.
Les gènes des protéines non structurelles, en revanche, codent pour des protéines, telles que la protéase similaire à la 3-chymotrypsine, la protéase similaire à la papaïne ou l'ARN polymérase ARN-dépendante, dont les fonctions régulent et dirigent les processus de réplication. assemblage de virus.
Vous trouverez ci-dessous une description des protéines structurelles individuelles, en mettant l'accent sur la protéine S, et des protéines non structurelles.
Pour en savoir plus : Coronavirus : qu'est-ce que c'est ?Pique Protéinée
Structure de la protéine de pointe SARS-CoV-2
La protéine de pointe (ou protéine S) du SRAS-CoV-2 (et de tous les coronavirus connus) recouvre la surface externe du virus, formant ces protubérances caractéristiques qui donnent au Nouveau Coronavirus l'apparence d'une couronne (dont il dérive, en fait, le terme « Coronavirus »).
La protéine de pointe pèse 180-200 kDa (lire kiloDalton) et est composée de 1.273 acides aminés.
Spike est composé de deux principaux composants d'acides aminés, appelés Sous-unité S1 (14-685) et Sous-unité S2 (686-1.273):
- La sous-unité S1 héberge une séquence d'acides aminés connue sous le nom de RBD (acronyme anglais qui signifie "Receptor Binding Domain", qui est le domaine de liaison au récepteur), qui est essentiel pour lier le virus aux cellules de l'hôte (c'est-à-dire l'être humain).
- La sous-unité S2, quant à elle, est le siège de séquences d'acides aminés (peptide de fusion, HR1, HR2, domaine transmembranaire et domaine cytoplasmique), dont la fonction finale est de favoriser la fusion et l'entrée du virus dans les cellules hôtes.
Dans son état natif (c'est-à-dire lorsque le virus n'infecte personne), la protéine de pointe se présente sous la forme de précurseur inactif. Cependant, lorsque le virus rencontre un organisme potentiel à infecter, il passe immédiatement à une forme active : les protéases des cellules cibles déclenchent le processus d'activation (c'est donc l'hôte lui-même qui l'active !), ce qui « casse » le pic. et forment les sous-unités S1 et S2.
Comment fonctionne la protéine de pointe SARS-CoV-2
Le fonctionnement de la protéine de pointe SARS-CoV-2 est complexe ; l'article en question vise à le simplifier au maximum, afin qu'il puisse être compris par les lecteurs.
La protéine de pointe est essentielle pour initier le processus d'infection de l'hôte ; en d'autres termes, c'est l'arme que le nouveau coronavirus utilise pour provoquer l'infection connue sous le nom de COVID-19.
Le processus d'infection induit par les pointes peut être divisé en deux étapes :
- Il lien à la cellule hôte. C'est la phase au cours de laquelle le virus attaque et se fixe aux cellules de l'organisme qu'il va ensuite infecter.
- La fusion de la membrane virale (essentiellement du virus) avec la membrane de la cellule hôte. C'est la phase qui permet au virus de pénétrer dans les cellules de l'organisme attaqué et d'y diffuser son génome.
Liaison aux cellules hôtes
La protéine de pointe se lie aux cellules hôtes via la séquence RBD de la sous-unité S1.
Des études scientifiques ont observé que la séquence RBD se lie aux cellules hôtes par une interaction avec le récepteur ACE2 placé à la surface de la membrane plasmique des cellules elles-mêmes.
L'ACE2 est une enzyme et est l'homologue de AS, la protéine responsable de la conversion de l'angiotensine 1-9.
Chez l'homme, l'ACE2 se trouve principalement à la surface de la membrane plasmique des cellules organiques telles que les poumons, les intestins, le cœur et les reins.
Une fois que la sous-unité S1 s'est liée à ACE2, la protéine S commence à changer de conformation; cet événement sert à favoriser la phase de fusion et l'entrée du virus dans la cellule hôte.
Le lien avec ACE2 et le changement conformationnel qui en résulte sont deux aspects fondamentaux pour la réalisation du Vaccin contre le SRAS-CoV-2 et comprendre les mécanismes de antigénicité et réponse immunitaire mis en œuvre par l'hébergeur.
Cependant, il y a un problème qui doit être considéré : des mutations dans la sous-unité S1 et, en particulier dans la séquence RBD, pourraient changer la façon dont le changement conformationnel se développe ; par conséquent, cela pourrait affecter les caractéristiques antigéniques et l'efficacité des vaccins (pour en savoir plus sur le sujet, nous vous recommandons de lire l'article consacré aux variants du SARS-CoV-2).
Fusion de cellules hôtes
La protéine de pointe fusionne le virus à la cellule hôte par l'intermédiaire des séquences d'acides aminés de la sous-unité S2.
Le processus de fusion virale a lieu sur la vague du changement de conformation de la protéine S induit par la liaison entre le RBD et le récepteur hôte ACE2 : le changement de conformation des pointes, en effet, rapproche la membrane virale de la membrane plasmique de la cellule hôte. , jusqu'à l'interaction, jusqu'à la fusion entre membranes et, enfin, jusqu'à l'incorporation du virus infectant.
Une fois que le génome viral est à l'intérieur de la cellule hôte, le virus commence sa réplication et le processus d'infection peut être considéré comme terminé.
Pour plus d'informations : Mutations de protéines de pointe : variantes du SARS-CoV-2Protéine du péricapside
Également appelées protéines E, les protéines péricapsides du SRAS-CoV-2 contribuent à la formation de la péricapside.
Ils constituent un groupe de très petites protéines, constituées uniquement de 75-109 acides aminés.
Malgré leur petite taille, les protéines E ont un rôle fonctionnel extrêmement important : en effet, elles soutiennent laassemblage et libération de virions.
En microbiologie, la particule virale mature est appelée virion, avec son acide nucléique (ADN ou ARN) enfermé dans une capsule protéique, appelée capside.
Des études à cet égard ont montré que la protéine E du SRAS-CoV-2 est l'une des viroporine, qui, une fois dans la cellule hôte, va se localiser sur la membrane de l'appareil de Golgi et du réticulum endoplasmique, pour faciliter l'assemblage et la libération des virions.
Une viroporine est une protéine virale qui agit comme un canal membranaire dans les cellules de l'hôte.
La protéine E du SRAS-CoV-2 est très similaire à celle du SRAS-CoV, bien qu'elle présente quelques différences par rapport à celle du MERS-CoV.
Protéine membranaire
Les protéines membranaires (ou protéines M) sont les protéines structurelles les plus abondantes dans le SRAS-CoV-2.
Ils ont une séquence d'acides aminés d'environ Articles 220.
La protéine M du SRAS-CoV-2 a plusieurs fonctions :
- Définit la forme de la péricapside;
- Interagissant avec les protéines E, N et S, il organise l'assemblage des virions.
La recherche a montré que sans la protéine M, mais avec toutes les autres protéines structurelles disponibles, le SARS-CoV-2 est incapable d'assembler de nouveaux virions au sein de l'hôte ; cela signifie que les protéines M jouent un rôle clé dans le processus susmentionné.
D'autre part, les preuves suggèrent que:
- L'interaction entre la protéine M et la protéine S assure l'incorporation de cette dernière dans les nouveaux virions ;
- L'interaction entre la protéine M et la protéine N stabilise la nucléocapside (c'est-à-dire le complexe ARN - protéine N) et favorise l'assemblage final des virions.
- Avec la protéine E, il contribue à la formation de la péricapside.
Protéine nucléocapside
La protéine N, ou protéine de nucléocapside, est la seule protéine du SRAS-CoV-2 capable de se lier au génome viral.
Sans surprise, grâce à cette propriété, il joue un rôle clé dans le processus de emballage d'ARN viral au sein des nouveaux virions.
Le complexe ARN viral - protéine N constitue ce qu'on appelle la nucléocapside.
Comme prévu, pour médier l'action de la protéine N est la protéine M : l'interaction entre ces deux protéines, en fait, stabilise la nucléocapside et favorise l'assemblage final des virions.
Il est à noter que les études sur la protéine N ont montré que cette dernière est également impliquée dans la transcription et la réplication des ARN viraux.
Suite à cette découverte, les experts ont commencé à considérer la protéine N comme une cible possible pour de nouveaux médicaments spécifiques au SRAS-CoV-2.
La protéine N est hautement conservée dans les coronavirus : par exemple, celle du SARS-CoV-2 a une séquence d'acides aminés superposable à 90 % à celle du SARS-CoV.
Protéines non structurelles
Origine et fonction des protéines non structurelles du SRAS-CoV-2
Le thème des protéines non structurales (en abrégé "nsp") du SARS-CoV-2 est quelque peu complexe.
Par conséquent, il a besoin d'une simplification, afin de le rendre plus facile à comprendre.
Premièrement, les protéines non structurelles du SARS-CoV-2 sont au total Articles 16.
Ils sont dérivés de deux grosses protéines, appelées polyprotéine 1a (pp1a) Et polyprotéine 1ab (pp1ab), qui à leur tour sont codés, respectivement, par les gènes viraux appelés répliques 1a e répliques 1ab.
Le processus de formation de protéines non structurales à partir des deux polyprotéines voit l'implication de deux enzymes virales spécifiques, appelées protéase et produit tôt par le virus; ces protéases traitent "tagliare" les polyprotéines en des points spécifiques, afin de donner naissance aux seules protéines non structurelles.
La stratégie des polyprotéines (dont sont dérivées des protéines plus petites) est très courante parmi les virus.
Il est intéressant de souligner qu'avant le processus de coupe, les protéines encore incluses dans les polyprotéines sont inactives, non fonctionnelles ; ils ne deviennent fonctionnels qu'après l'intervention des protéases et leur clivage par rapport aux chaînes d'acides aminés majeurs.
La fonction principale des protéines non structurelles du SARS-CoV-2 est de traiter transcription et réplication ARN viral.
Cependant, il convient de noter que ces protéines sont également impliquées dans pathogenèse virale.
Protéase SARS-CoV-2
Deux protéines non structurelles fondamentales pour le SARS-CoV-2 sont, sans aucun doute, les protéases qui s'occupent de "couper" les polyprotéines et de former les protéines utiles pour la transcription et la réplication de l'ARN viral.
Ces protéases sont appelées protéase semblable à 3-chimotripsine (abrégé en 3CLpro) Et protéase de type papaïne (abrégé en PLpro).
Considérant que les protéines auxquelles elles donnent naissance servent ensuite à propager l'infection chez l'hôte, les protéases en question représentent une cible pharmacologique intéressante.
ARN ARN polymérase dépendante
L'ARN polymérase ARN-dépendante c'est la protéine non structurale du SARS-CoV-2 indispensable à la réplication du génome viral destiné aux nouveaux virions.
Cette protéine non structurale représenterait également une cible pharmacologique intéressante.
Pathogénie virale
Comment le SRAS-CoV-2 provoque l'infection au COVID-19
Le SARS-CoV-2 commence le processus infectieux lorsque, grâce à la protéine de pointe, il parvient à envahir les cellules de l'hôte.
Comme décrit dans le chapitre consacré à la protéine S, l'interaction entre la séquence de pointe RBD et le récepteur ACE2 présent sur la membrane plasmique des cellules des voies respiratoires de l'hôte assure lui-même l'entrée du virus dans l'organisme hôte.
Dès son entrée, le SARS-CoV-2 « s'approprie » les ribosomes de l'hôte et les exploite pour traduire son propre génome en ARN et créer les protéines nécessaires à la réplication du même matériel génétique et à l'assemblage de nouveaux virions.
Sur la base de ce qui précède, un rôle clé dans la transcription et la réplication de l'ARN viral appartient aux protéines non structurelles.
Avec la transcription et la réplication du génome viral, le SRAS-CoV-2 commence à se propager dans l'hôte, déclenchant la véritable maladie infectieuse.
Dans cette phase, le virus agit sur l'organisme hôte à la fois avec unactivité cytocide (c'est-à-dire qu'il tue les cellules) avec les deux mécanismes à médiation immunitaire.
En ce qui concerne l'activité cytocide, les preuves suggèrent que le SRAS-CoV-2 induit apoptose (mort cellulaire) e lyse cellulaire; plus en détail, il est apparu que le virus produit des syncytia au sein de la cellule infectée et provoque la rupture de l'appareil de Golgi après réplication.
En ce qui concerne les mécanismes à médiation immunitaire, la recherche a montré que le SRAS-CoV-2 implique à la fois le système immunitaire inné, être cela adaptatif (anticorps et lymphocytes T).
Pourquoi le SARS-CoV-2 est-il plus infectieux que le coronavirus du SRAS ?
Le SRAS-CoV, le coronavirus responsable du SRAS, envahit également les cellules hôtes en exploitant l'interaction entre le RBD et le récepteur ACE2 présent sur les cellules des voies respiratoires.
Cependant, il existe une différence importante entre ce type de lien et celui mis en place par le SARS-CoV-2 : la séquence RBD du Coronavirus responsable du COVID-19 a beaucoup plus d'affinité pour ACE2 et s'y lie beaucoup plus efficacement, résultant beaucoup plus efficace dans le processus d'invasion des cellules hôtes.
Des études scientifiques à cet égard ont montré que la différence d'interaction décrite ci-dessus est due à un composition différente en acides aminés entre le RBD du SARS-CoV et le RBD du SARS-CoV-2 ; en particulier, il existe deux régions d'acides aminés avec des différences importantes.
Cette différence d'affinité explique plusieurs aspects :
- La raison pour laquelle le SARS-CoV-2 a un R0 plus élevé que le SARS-CoV ;
- La raison pour laquelle les médicaments et les vaccins qui ciblaient la séquence RBD du SRAS-CoV et semblaient être efficaces ne conviennent pas contre le SARS-CoV-2.
Réaction inflammatoire
SARS-CoV-2 et libération de cytokines
À l'entrée du SRAS-CoV-2, le système immunitaire de l'organisme hôte infecté est activé.
À ce stade, des éléments du système immunitaire (par exemple les lymphocytes T) atteignent le site de l'infection et attaquent le virus.
Chez la plupart des gens, le processus ci-dessus réussit, éliminant le virus du corps et permettant à l'individu de se rétablir.
Dans un certain pourcentage de cas cependant, il arrive que la maladie infectieuse prenne des connotations plus graves et que le SARS-CoV-2 stimule une réponse immunitaire aberrant.
Dans ce cas, dans les situations susmentionnées, il est apparu que le virus provoque une surproduction de cytokines pro-inflammatoires (ex : interleukine-1, interleukine-2, interleukine-6 etc.) qui s'accumulent dans les poumons, au point d'endommager le parenchyme pulmonaire.
Les cytokines pro-inflammatoires résultent de l'activité de certaines cellules du système immunitaire.
Dans des conditions normales, ils servent à réguler la réponse immunitaire, l'inflammation et l'hématopoïèse.
De plus, des données cliniques et d'autres recherches ont montré que la surproduction de cytokines pro-inflammatoires observée en présence d'une infection sévère par le SRAS-CoV-2 peut s'étendre jusqu'à autres organes (es: cuore), provoquant la dysfonctionnement, et avoir des répercussions sur processus de coagulation, induisant la formation de thrombus.
Lorsque le SARS-CoV-2 déclenche une surproduction extensive de cytokines pro-inflammatoires, les experts définissent le phénomène avec l'expression "syndrome de tempête de cytokines".
Bibliographie
- Ahmad Abu Turab Naqvi, Kisa Fatima, Taj Mohammad, Urooj Fatima, Indrakant K. Singh, Archana Singh, Shaikh Muhammad Atif, Gururao Hariprasad, Gulam Mustafa Hasan et Md. Imtaiyaz Hassan (101 octobre 2021) Aperçu du SRAS-CoV-2 génome, structure, évolution, pathogenèse et thérapies : Approche de la génomique structurale. Biochim Biophys Acta Mol Basis Dis.; 1866(10) : 165878.
- Yuan Huang, Chan Yang, Xin-feng Xu, Wei Xu et Shu-wen Liu (03 août 2021) Propriétés structurelles et fonctionnelles de la protéine de pointe SARS-CoV-2 : développement potentiel de médicaments antivirus pour COVID-19. Acta Pharmacologica Sinica volume 41, pages 1141-1149.
- Muge Cevik, conférencier clinicien, Krutika Kuppalli, Jason Kindrachuk et Malik Peiris (23 octobre 2021) Virologie, transmission et pathogenèse du SRAS-CoV-2. BMJ ; 371 : m3862.