Introducción
Desde hace más de un año, la atención del mundo se ha dirigido a COVID-19, infección respiratoria causada por el nuevo Coronavirus SARS-CoV-2, Y el pandemia que resultó de ella.
Los datos epidemiológicos del momento dicen que: El SARS-CoV-2 está presente en más de 200 países del mundo, aproximadamente 19 millones de personas han enfermado por COVID-113 en todo el mundo (febrero de 2021) y, de estos, no 2,5 millones murió.
El SARS-CoV-2 es un virus que afecta principalmente al tracto respiratorio, provocando síntomas como tos, resfriado, fiebre y, en los casos más graves, dificultad para respirar; a veces, sin embargo, también puede inducir inflamación sistémica, provocando sepsis, insuficiencia cardíaca y disfunción multiorgánica.
La infección por SARS-CoV-2 es particularmente peligrosa para las personas mayores de 60 años, para las personas con enfermedades crónicas (por ejemplo: diabetes, enfermedad coronaria) y para las personas en terapia con medicamentos depresores del sistema inmunológico (por ejemplo: quimioterapia, inmunosupresores).
Este artículo tiene como objetivo analizar la estructura, el genoma y las proteínas del SARS-CoV-2, y proporcionar información fundamental relacionada con la patogénesis del virus.
Para más información: SARS-CoV-2: Cómo reconocer los primeros síntomas y qué hacerEstructura del SARS-CoV-2
Cómo se elabora el SARS-CoV-2: estructura, genoma y proteínas
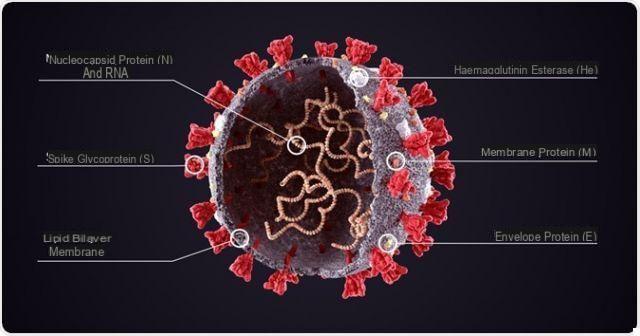
Ejemplo de beta-coronavirus, SARS-CoV-2 es un virus de ARN monocatenario positivo, equipado con pericapsida (la envoltura).
El pericapsid es una especie de envoltura que se coloca alrededor de la cápside de algunos virus; está formado por fosfolípidos y glicoproteínas.
El SARS-CoV-2 posee un genoma de 29.881 bases nitrogenadas, que codifica para 9.860 aminoácidos.
Este genoma se descompone en genes para proteínas estructurales y genes para proteínas no estructurales.
Los genes de proteínas estructurales codifican pico de proteínas (abreviado como S), la proteina del pericapsida (abreviado a E, de sobre), el proteína de membrana (abreviado como M) y el proteína de la nucleocápside (abreviado como N).
Como sugiere el nombre, las proteínas estructurales se combinan para formar la estructura del SARS-CoV-2.
Los genes para proteínas no estructurales, por su parte, codifican proteínas, como la proteasa similar a la 3-quimotripsina, la proteasa similar a la papaína o la ARN polimerasa dependiente de ARN, cuyas funciones son regular y dirigir los procesos de replicación. ensamblaje de virus.
A continuación se muestra una descripción de las proteínas estructurales individuales, con especial atención a la proteína S, y de las proteínas no estructurales.
Para obtener más información: Coronavirus: ¿Qué son?Pico de proteína
Estructura de la proteína espiga del SARS-CoV-2
La proteína pico (o proteína S) del SARS-CoV-2 (y de todos los Coronavirus conocidos) recubre la superficie externa del virus, formando esas protuberancias características que le dan al Nuevo Coronavirus la apariencia de una corona (de la cual deriva, en de hecho, el término "Coronavirus").
La proteína de pico pesa 180-200 kDa (leemos kiloDalton) y está compuesta de 1.273 aminoácidos.
Spike se compone de dos componentes principales de aminoácidos, llamados Subunidad S1 (14-685) y Subunidad S2 (686, 1.273):
- La subunidad S1 alberga una secuencia de aminoácidos conocida como RBD (Acrónimo en inglés de "Receptor Binding Domain"), que es esencial para unir el virus a las células del huésped (es decir, el ser humano).
- La subunidad S2, por su parte, es el sitio de secuencias de aminoácidos (péptido de fusión, HR1, HR2, dominio transmembrana y dominio citoplasmático), cuya función final es favorecer la fusión y entrada del virus en las células hospedadoras.
En su estado nativo (es decir, cuando el virus no está infectando a nadie), la proteína de pico está en forma de precursor inactivo. Sin embargo, cuando el virus se encuentra con un organismo potencial para ser infectado, cambia inmediatamente a una forma activa: las proteasas de las células diana desencadenan el proceso de activación (¡por lo que es el propio anfitrión quien lo activa!), Que "rompen" el pico. y forman las subunidades S1 y S2.
Cómo funciona la proteína espiga del SARS-CoV-2
El funcionamiento de la proteína espiga del SARS-CoV-2 es complejo; el artículo en cuestión tiene como objetivo simplificarlo tanto como sea posible, para que pueda ser entendido por los lectores.
La proteína de pico es esencial para iniciar el proceso de infección del huésped; es decir, es el arma que utiliza el Nuevo Coronavirus para provocar la infección conocida como COVID-19.
El proceso de infección impulsado por picos se puede dividir en dos etapas:
- Il enlace a la célula huésped. Es la fase en la que el virus ataca y se une a las células del organismo que luego infectará.
- La fusión de la membrana viral (esencialmente del virus) con la membrana de la célula huésped. Es la fase que permite que el virus ingrese a las células del organismo atacado y disemine allí su genoma.
Unión a las células huésped
La proteína espiga se une a las células huésped a través de la secuencia RBD de la subunidad S1.
Estudios científicos han observado que la secuencia RBD se une a las células huésped mediante una interacción con el Receptor ACE2 colocado en la superficie de la membrana plasmática de las propias células.
ACE2 es una enzima y es homólogo de ACE, la proteina deputata a convertire l'angiotensina 1-9.
En los seres humanos, la ECA2 se encuentra principalmente en la superficie de la membrana plasmática de las células de órganos como los pulmones, los intestinos, el corazón y los riñones.
Una vez que la subunidad S1 se ha unido a ACE2, la proteína S comienza a cambiar conformación; este evento sirve para favorecer la fase de fusión y la entrada del virus en la célula huésped.
El vínculo con ACE2 y el cambio conformacional resultante son dos aspectos fundamentales para la realización del Vacuna contra el SARS-CoV-2 y comprender los mecanismos de antigenicità y respuesta inmune implementado por el anfitrión.
Sin embargo, existe un problema que debe ser considerado: mutaciones en la subunidad S1 y, en particular, en la secuencia RBD, podrían cambiar la forma en que se desarrolla el cambio conformacional; en consecuencia, esto podría tener un impacto en las características antigénicas y la eficacia de las vacunas (para conocer más sobre el tema, recomendamos leer el artículo dedicado a las variantes del SARS-CoV-2).
Fusión de la célula huésped
La proteína espiga fusiona el virus con la célula huésped a través de las secuencias de aminoácidos de la subunidad S2.
El proceso de fusión del virus tiene lugar en la onda del cambio conformacional de la proteína S inducida por el enlace entre el RBD y el receptor ACE2 del huésped: el cambio en la conformación de la espiga, de hecho, acerca la membrana viral a la membrana plasmática de la célula huésped. , hasta la interacción, la fusión entre membranas y, finalmente, la incorporación del virus infectante.
Una vez que el genoma viral está dentro de la célula huésped, el virus comienza su replicación y el proceso de infección puede considerarse completo.
Para obtener más información: Mutaciones de proteínas de pico: variantes del SARS-CoV-2Proteina del Pericapside
También conocidas como proteínas E, las proteínas del pericapsido del SARS-CoV-2 contribuyen a la formación del pericapsido.
Constituyen un grupo de proteínas muy pequeñas, que consta únicamente de 75-109 aminoácidos.
A pesar de su pequeño tamaño, las proteínas E tienen un papel funcional extremadamente importante: de hecho, apoyan laensamblaje y liberación de viriones.
En microbiología, la partícula viral madura se llama virión, con su ácido nucleico (ADN o ARN) encerrado en una cápsula de proteína, llamada cápside.
Los estudios a este respecto han demostrado que la proteína E del SARS-CoV-2 es una viroporina, que, una vez en la célula huésped, se va a localizar en la membrana del aparato de Golgi y del retículo endoplásmico, para facilitar el ensamblaje y liberación de viriones.
Una viroporina es una proteína viral que actúa como un canal de membrana dentro de las células del huésped.
La proteína E del SARS-CoV-2 es muy similar a la del SARS-CoV, aunque tiene algunas diferencias con la del MERS-CoV.
Proteína de membrana
Las proteínas de membrana (o proteínas M) son las proteínas estructurales más abundantes en el SARS-CoV-2.
Tienen una secuencia de aminoácidos de aproximadamente 220 artículos.
La proteína M del SARS-CoV-2 tiene varias funciones:
- Define la forma del pericapsid;
- Interactuando con las proteínas E, N y S, organiza el ensamblaje de viriones.
La investigación ha demostrado que sin la proteína M, pero con todas las demás proteínas estructurales disponibles, el SARS-CoV-2 no puede ensamblar nuevos viriones dentro del huésped; esto significa que las proteínas M juegan un papel clave en el proceso antes mencionado.
Por otro lado, la evidencia sugiere que:
- La interacción entre la proteína M y la proteína S asegura la incorporación de esta última en los nuevos viriones;
- La interacción entre la proteína M y la proteína N estabiliza la nucleocápside (es decir, el complejo ARN-proteína N) y promueve el ensamblaje final de los viriones.
- Junto con la proteína E, contribuye a la formación del pericápsido.
Proteína nucleocápsida
La proteína N, o proteína de la nucleocápsida, es la única proteína del SARS-CoV-2 capaz de unirse al genoma viral.
No en vano, gracias a esta propiedad, juega un papel clave en el proceso de envasado de ARN viral dentro de los nuevos viriones.
El complejo proteico ARN-N viral constituye la denominada nucleocápside.
Como se anticipó, para mediar la acción de la proteína N está la proteína M: la interacción entre estas dos proteínas, de hecho, estabiliza la nucleocápside y promueve el ensamblaje final de los viriones.
Cabe señalar que los estudios sobre la proteína N mostraron que esta última también participa en la transcripción y replicación del ARN viral.
Tras este descubrimiento, los expertos empezaron a considerar la proteína N como un posible objetivo de nuevos fármacos específicos para el SARS-CoV-2.
La proteína N está altamente conservada en los coronavirus: por ejemplo, la del SARS-CoV-2 tiene una secuencia de aminoácidos que es 90% superponible a la del SARS-CoV.
Proteínas no estructurales
Origen y función de las proteínas no estructurales del SARS-CoV-2
El tema de las proteínas no estructurales (abreviado como "nsp") de SARS-CoV-2 es algo complejo.
Por tanto, necesita una simplificación, para que sea más fácil de entender.
Primero, las proteínas no estructurales del SARS-CoV-2 están en total 16 artículos.
Se derivan de dos proteínas grandes, llamadas poliproteina 1a (pp1a) Y poliproteína 1ab (pp1ab), que a su vez están codificados, respectivamente, por los genes virales conocidos como replicasi 1a e replicasi 1ab.
El proceso de formación de proteínas no estructurales a partir de las dos poliproteínas ve la participación de dos enzimas virales específicas, llamadas proteasa y producido temprano por el virus; estas proteasas tratan "tagliare" le poliproteína en puntos específicos, para dar lugar a las proteínas no estructurales únicas.
La estrategia de poliproteínas (de la que se derivan proteínas más pequeñas) es muy común entre los virus.
Es interesante señalar que, antes del proceso de corte, las proteínas aún incluidas en las poliproteínas están inactivas, no funcionales; se vuelven funcionales sólo después de la intervención de las proteasas y su escisión con respecto a las principales cadenas de aminoácidos.
La función principal de las proteínas no estructurales del SARS-CoV-2 es tratar transcripción y replicación ARN viral.
Sin embargo, cabe señalar que estas proteínas también están involucradas en patogenia viral.
Proteasa del SARS-CoV-2
Dos proteínas no estructurales fundamentales para el SARS-CoV-2 son, sin duda, las proteasas que se ocupan de "cortar" las poliproteínas y formar las proteínas útiles para la transcripción y replicación del ARN viral.
Estas proteasas se conocen como proteasa similar a 3-chimotripsina (abreviado a 3CLpro) Y proteasa similar a la papaína (abreviado a PLpro).
Teniendo en cuenta que las proteínas a las que dan lugar sirven para propagar la infección en el huésped, las proteasas en cuestión representan una interesante diana farmacológica.
ARN polimerasa dependiente de ARN
L'ARN polimerasa dependiente de ARN es la proteína no estructural del SARS-CoV-2 esencial para la replicación del genoma viral destinado a los nuevos viriones.
Esta proteína no estructural también representaría un atractivo objetivo farmacológico.
Patogenia viral
Cómo el SARS-CoV-2 causa la infección por COVID-19
El SARS-CoV-2 inicia el proceso infeccioso cuando, a través de la proteína de pico, logra invadir las células del huésped.
Como se describe en el capítulo dedicado a la proteína S, la interacción entre la secuencia de picos de RBD y el receptor ACE2 presente en la membrana plasmática de las células del tracto respiratorio del propio huésped asegura la entrada del virus en el organismo huésped.
Al entrar, el SARS-CoV-2 "se apropia" de los ribosomas del huésped y los explota para traducir su propio genoma en ARN y crear las proteínas necesarias para la replicación del mismo material genético y el ensamblaje de nuevos viriones.
En base a lo anterior, un papel clave en la transcripción y replicación del ARN viral pertenece a las proteínas no estructurales.
Con la transcripción y replicación del genoma viral, el SARS-CoV-2 comienza a diseminarse en el huésped, iniciando la enfermedad infecciosa real.
En esta fase, el virus actúa sobre el organismo huésped tanto con unactividad citocida (es decir, mata células) con ambos mecanismos inmunomediados.
Con respecto a la actividad citocida, la evidencia sugiere que el SARS-CoV-2 induce apoptosis (muerte celular) e lisis celular; más en detalle, se descubrió que el virus produce sincitios dentro de la célula infectada y hace que el aparato de Golgi se rompa después de la replicación.
En cuanto a los mecanismos inmunomediados, la investigación ha demostrado que el SARS-CoV-2 involucra tanto a la Sistema inmune innato, sé eso adaptado (anticuerpos y linfocitos T).
¿Por qué el SARS-CoV-2 es más infeccioso que el coronavirus del SARS?
El SARS-CoV, el coronavirus responsable del SARS, también invade las células huésped aprovechando la interacción entre el RBD y el receptor ACE2 presente en las células del tracto respiratorio.
Sin embargo, existe una diferencia importante entre este tipo de vínculo y el establecido por el SARS-CoV-2: la secuencia RBD del Coronavirus responsable del COVID-19 ha mucha más afinidad para ACE2 y se une a él mucho más eficientemente, resultando mucho más eficaz en el proceso de invasión de las células huésped.
Los estudios científicos a este respecto han demostrado que la diferencia en la interacción descrita anteriormente se debe a una diferente composición de aminoácidos entre RBD de SARS-CoV y RBD de SARS-CoV-2; en particular, hay dos regiones de aminoácidos con diferencias importantes.
Esta diferencia de afinidad explica varios aspectos:
- Por qué el SARS-CoV-2 tiene un R0 más alto que el SARS-CoV;
- La razón por la que los medicamentos y vacunas que se dirigieron a la secuencia RBD del SARS-CoV y parecieron ser efectivos no son adecuados contra el SARS-CoV-2.
Respuesta inflamatoria
Liberación de SARS-CoV-2 y citocinas
Tras la entrada de SARS-CoV-2, se activa el sistema inmunológico del organismo huésped infectado.
En este punto, los elementos del sistema inmunológico (por ejemplo, los linfocitos T) llegan al sitio de la infección y atacan al virus.
En la mayoría de las personas, el proceso anterior es exitoso, elimina el virus del cuerpo y permite que la persona se recupere.
En un cierto porcentaje de casos, sin embargo, sucede que la enfermedad infecciosa adquiere connotaciones más graves y que el SARS-CoV-2 estimula una respuesta inmune. aberrante.
En este caso, en las situaciones antes mencionadas, surgió que el virus provoca una sobreproducción de citocinas proinflamatorias (por ejemplo: interleucina-1, interleucina-2, interleucina-6, etc.) que se acumulan en los pulmones, hasta el punto de dañar el parénquima pulmonar.
Las citocinas proinflamatorias surgen de la actividad de ciertas células del sistema inmunológico.
En condiciones normales, sirven para regular la respuesta inmunitaria, la inflamación y la hematopoyesis.
Además, los datos clínicos y otras investigaciones han demostrado que la sobreproducción de citocinas proinflamatorias observada en presencia de una infección grave por SARS-CoV-2 puede extenderse a otros órganos (ej .: corazón), causando el disfuncióny tienen repercusiones en procesos de coagulación, induciendo la formación de trombo.
Cuando el SARS-CoV-2 desencadena una extensa sobreproducción de citocinas proinflamatorias, los expertos definen el fenómeno con la expresión "síndrome de tormenta de citocinas".
Bibliografía
- Ahmad Abu Turab Naqvi, Kisa Fatima, Taj Mohammad, Urooj Fatima, Indrakant K. Singh, Archana Singh, Shaikh Muhammad Atif, Gururao Hariprasad, Gulam Mustafa Hasan y Md. Imtaiyaz Hassan (101 de octubre de 2021) Perspectivas sobre el SARS-CoV-2 genoma, estructura, evolución, patogenia y terapias: enfoque de la genómica estructural. Biochim Biophys Acta Mol Basis Dis .; 1866 (10): 165878.
- Yuan Huang, Chan Yang, Xin-feng Xu, Wei Xu y Shu-wen Liu (03 de agosto de 2021) Propiedades estructurales y funcionales de la proteína pico del SARS-CoV-2: desarrollo potencial de fármacos antivirus para COVID-19. Acta Pharmacologica Sinica volumen 41, páginas 1141–1149.
- Muge Cevik, conferencista clínico, Krutika Kuppalli, Jason Kindrachuk y Malik Peiris (23 de octubre de 2021) Virología, transmisión y patogenia del SARS-CoV-2. BMJ; 371: m3862.